Numpy Basic
1 Numpy 기본
도구 - 넘파이(NumPy)
- 넘파이(NumPy)는 파이썬의 과학 컴퓨팅을 위한 기본 라이브러리입니다.
- 넘파이의 핵심은 강력한 N-차원 배열 객체입니다.
- 선형 대수, 푸리에(Fourier) 변환, 유사 난수 생성과 같은 유용한 함수들도 제공합니다.
 Jupyter에서 실행하기 Jupyter에서 실행하기
|
 구글 코랩에서 실행하기 구글 코랩에서 실행하기
|
2 배열 생성
numpy를 임포트해 보죠. 대부분의 사람들이 np로 알리아싱하여 임포트합니다:
import numpy as np2.1 np.zeros
zeros 함수는 0으로 채워진 배열을 만듭니다:
np.zeros(5)array([0., 0., 0., 0., 0.])2D 배열(즉, 행렬)을 만들려면 원하는 행과 열의 크기를 튜플로 전달합니다. 예를 들어 다음은 \(3 \times 4\) 크기의 행렬입니다:
np.zeros((3,4))array([[0., 0., 0., 0.],
[0., 0., 0., 0.],
[0., 0., 0., 0.]])3 용어
- 넘파이에서 각 차원을 축(axis) 이라고 합니다
- 축의 개수를 랭크(rank) 라고 합니다.
- 예를 들어, 위의 \(3 \times 4\) 행렬은 랭크 2인 배열입니다(즉 2차원입니다).
- 첫 번째 축의 길이는 3이고 두 번째 축의 길이는 4입니다.
- 배열의 축 길이를 배열의 크기(shape)라고 합니다.
- 예를 들어, 위 행렬의 크기는
(3, 4)입니다. - 랭크는 크기의 길이와 같습니다.
- 예를 들어, 위 행렬의 크기는
- 배열의 사이즈(size)는 전체 원소의 개수입니다. 축의 길이를 모두 곱해서 구할 수 있습니다(가령, \(3 \times 4=12\)).
a = np.zeros((3,4))
aarray([[0., 0., 0., 0.],
[0., 0., 0., 0.],
[0., 0., 0., 0.]])a.shape(3, 4)a.ndim # len(a.shape)와 같습니다2a.size124 N-차원 배열
임의의 랭크 수를 가진 N-차원 배열을 만들 수 있습니다. 예를 들어, 다음은 크기가 (2,3,4)인 3D 배열(랭크=3)입니다:
np.zeros((2,3,4))array([[[0., 0., 0., 0.],
[0., 0., 0., 0.],
[0., 0., 0., 0.]],
[[0., 0., 0., 0.],
[0., 0., 0., 0.],
[0., 0., 0., 0.]]])5 배열 타입
넘파이 배열의 타입은 ndarray입니다:
type(np.zeros((3,4)))numpy.ndarray5.1 np.ones
ndarray를 만들 수 있는 넘파이 함수가 많습니다.
다음은 1로 채워진 \(3 \times 4\) 크기의 행렬입니다:
np.ones((3,4))array([[1., 1., 1., 1.],
[1., 1., 1., 1.],
[1., 1., 1., 1.]])5.2 np.full
주어진 값으로 지정된 크기의 배열을 초기화합니다. 다음은 π로 채워진 \(3 \times 4\) 크기의 행렬입니다.
np.full((3,4), np.pi)array([[3.14159265, 3.14159265, 3.14159265, 3.14159265],
[3.14159265, 3.14159265, 3.14159265, 3.14159265],
[3.14159265, 3.14159265, 3.14159265, 3.14159265]])5.3 np.empty
초기화되지 않은 \(2 \times 3\) 크기의 배열을 만듭니다(배열의 내용은 예측이 불가능하며 메모리 상황에 따라 달라집니다):
np.empty((2,3))array([[0., 0., 0.],
[0., 0., 0.]])5.4 np.array
array 함수는 파이썬 리스트를 사용하여 ndarray를 초기화합니다:
np.array([[1,2,3,4], [10, 20, 30, 40],[3,4,5,6]])array([[ 1, 2, 3, 4],
[10, 20, 30, 40],
[ 3, 4, 5, 6]])5.5 np.arange
파이썬의 기본 range 함수와 비슷한 넘파이 arange 함수를 사용하여 ndarray를 만들 수 있습니다:
np.arange(1, 5)array([1, 2, 3, 4])부동 소수도 가능합니다:
np.arange(1.0, 5.0)array([1., 2., 3., 4.])파이썬의 기본 range 함수처럼 건너 뛰는 정도를 지정할 수 있습니다:
np.arange(1, 5, 0.5)array([1. , 1.5, 2. , 2.5, 3. , 3.5, 4. , 4.5])부동 소수를 사용하면 원소의 개수가 일정하지 않을 수 있습니다. 예를 들면 다음과 같습니다:
print(np.arange(0, 5/3, 1/3)) # 부동 소수 오차 때문에, 최댓값은 4/3 또는 5/3이 됩니다.
print(np.arange(0, 5/3, 0.333333333))
print(np.arange(0, 5/3, 0.333333334))[0. 0.33333333 0.66666667 1. 1.33333333 1.66666667]
[0. 0.33333333 0.66666667 1. 1.33333333 1.66666667]
[0. 0.33333333 0.66666667 1. 1.33333334]5.6 np.linspace
이런 이유로 부동 소수를 사용할 땐 arange 대신에 linspace 함수를 사용하는 것이 좋습니다. linspace 함수는 지정된 개수만큼 두 값 사이를 나눈 배열을 반환합니다(arange와는 다르게 최댓값이 포함됩니다):
print(np.linspace(0, 5/3, 6)) #n=6[0. 0.33333333 0.66666667 1. 1.33333333 1.66666667]5.7 np.rand & np.randn
넘파이의 random 모듈에는 ndarray를 랜덤한 값으로 초기화할 수 있는 함수들이 많이 있습니다. 예를 들어, 다음은 (균등 분포인) 0과 1사이의 랜덤한 부동 소수로 \(3 \times 4\) 행렬을 초기화합니다:
np.random.rand(3,4)array([[0.10446653, 0.08608099, 0.39113434, 0.51368828],
[0.5422316 , 0.60730771, 0.14296635, 0.65012684],
[0.32191595, 0.67644108, 0.97250068, 0.02085189]])다음은 평균이 0이고 분산이 1인 일변량 정규 분포(가우시안 분포)에서 샘플링한 랜덤한 부동 소수를 담은 \(3 \times 4\) 행렬입니다:
np.random.randn(3,4)array([[-8.45928911e-01, -2.54018182e-04, 1.58171732e-01,
-1.56553088e-01],
[ 1.26984307e+00, 1.06076160e-01, 1.79499825e+00,
-1.34570830e-01],
[ 1.47976512e+00, 3.92371306e-01, 1.82902549e+00,
1.14404415e+00]])이 분포의 모양을 알려면 맷플롯립을 사용해 그려보는 것이 좋습니다(더 자세한 것은 맷플롯립 튜토리얼을 참고하세요):
%matplotlib inline
import matplotlib.pyplot as pltplt.hist(np.random.rand(100000), density=True, bins=100, histtype="step", color="blue", label="rand")
plt.hist(np.random.randn(100000), density=True, bins=100, histtype="step", color="red", label="randn")
plt.axis([-2.5, 2.5, 0, 1.1])
plt.legend(loc = "upper left")
plt.title("Random distributions")
plt.xlabel("Value")
plt.ylabel("Density")
plt.show()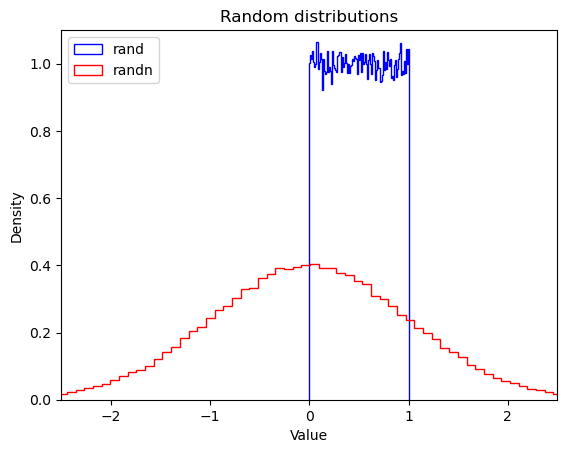
5.8 np.fromfunction
함수를 사용하여 ndarray를 초기화할 수도 있습니다:
def my_function(z, y, x):
return x + 10 * y + 100 * z
np.fromfunction(my_function, (3, 2, 10))array([[[ 0., 1., 2., 3., 4., 5., 6., 7., 8., 9.],
[ 10., 11., 12., 13., 14., 15., 16., 17., 18., 19.]],
[[100., 101., 102., 103., 104., 105., 106., 107., 108., 109.],
[110., 111., 112., 113., 114., 115., 116., 117., 118., 119.]],
[[200., 201., 202., 203., 204., 205., 206., 207., 208., 209.],
[210., 211., 212., 213., 214., 215., 216., 217., 218., 219.]]])넘파이는 먼저 크기가 (3, 2, 10)인 세 개의 ndarray(차원마다 하나씩)를 만듭니다. 각 배열은 축을 따라 좌표 값과 같은 값을 가집니다. 예를 들어, z 축에 있는 배열의 모든 원소는 z-축의 값과 같습니다:
[[[ 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.]
[ 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.]]
[[ 1. 1. 1. 1. 1. 1. 1. 1. 1. 1.]
[ 1. 1. 1. 1. 1. 1. 1. 1. 1. 1.]]
[[ 2. 2. 2. 2. 2. 2. 2. 2. 2. 2.]
[ 2. 2. 2. 2. 2. 2. 2. 2. 2. 2.]]]위의 식 x + 10 * y + 100 * z에서 x, y, z는 사실 ndarray입니다(배열의 산술 연산에 대해서는 아래에서 설명합니다). 중요한 점은 함수 my_function이 원소마다 호출되는 것이 아니고 딱 한 번 호출된다는 점입니다. 그래서 매우 효율적으로 초기화할 수 있습니다.
6 배열 데이터
6.1 dtype
넘파이의 ndarray는 모든 원소가 동일한 타입(보통 숫자)을 가지기 때문에 효율적입니다. dtype 속성으로 쉽게 데이터 타입을 확인할 수 있습니다:
c = np.arange(1, 5)
print(c.dtype, c)int32 [1 2 3 4]c = np.arange(1.0, 5.0)
print(c.dtype, c)float64 [1. 2. 3. 4.]넘파이가 데이터 타입을 결정하도록 내버려 두는 대신 dtype 매개변수를 사용해서 배열을 만들 때 명시적으로 지정할 수 있습니다:
d = np.arange(1, 5, dtype=np.complex64)
print(d.dtype, d)complex64 [1.+0.j 2.+0.j 3.+0.j 4.+0.j]가능한 데이터 타입은 int8, int16, int32, int64, uint8|16|32|64, float16|32|64, complex64|128가 있습니다. 전체 리스트는 온라인 문서를 참고하세요.
6.2 itemsize
itemsize 속성은 각 아이템의 크기(바이트)를 반환합니다:
e = np.arange(1, 5, dtype=np.complex64)
print(e)
print(e.itemsize)[1.+0.j 2.+0.j 3.+0.j 4.+0.j]
86.3 data 버퍼
배열의 데이터는 1차원 바이트 버퍼로 메모리에 저장됩니다. data 속성을 사용해 참조할 수 있습니다(사용할 일은 거의 없겠지만요).
f = np.array([[1,2],[1000, 2000]], dtype=np.int32)
print(f)
print(f.data)[[ 1 2]
[1000 2000]]
<memory at 0x000002664FD6BEE0>파이썬 2에서는 f.data가 버퍼이고 파이썬 3에서는 memoryview입니다.
if (hasattr(f.data, "tobytes")):
data_bytes = f.data.tobytes() # python 3
else:
data_bytes = memoryview(f.data).tobytes() # python 2
data_bytesb'\x01\x00\x00\x00\x02\x00\x00\x00\xe8\x03\x00\x00\xd0\x07\x00\x00'여러 개의 ndarray가 데이터 버퍼를 공유할 수 있습니다. 하나를 수정하면 다른 것도 바뀝니다. 잠시 후에 예를 살펴 보겠습니다.
7 배열 크기 변경
7.1 자신을 변경
ndarray의 shape 속성을 지정하면 간단히 크기를 바꿀 수 있습니다. 배열의 원소 개수는 동일하게 유지됩니다.
g = np.arange(24)
print(g)
print("랭크:", g.ndim)[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23]
랭크: 1g.shape = (6, 4)
print(g)
print("랭크:", g.ndim)[[ 0 1 2 3]
[ 4 5 6 7]
[ 8 9 10 11]
[12 13 14 15]
[16 17 18 19]
[20 21 22 23]]
랭크: 2g.shape = (2, 3, 4)
print(g)
print("랭크:", g.ndim)[[[ 0 1 2 3]
[ 4 5 6 7]
[ 8 9 10 11]]
[[12 13 14 15]
[16 17 18 19]
[20 21 22 23]]]
랭크: 3g[1,1,1] # 3차원 인덱싱 0,1 ~ 순177.2 reshape
reshape 함수는 동일한 데이터를 가리키는 새로운 ndarray 객체를 반환합니다. 한 배열을 수정하면 다른 것도 함께 바뀝니다.
g2 = g.reshape(4,6)
print(g2)
print("랭크:", g2.ndim)[[ 0 1 2 3 4 5]
[ 6 7 8 9 10 11]
[12 13 14 15 16 17]
[18 19 20 21 22 23]]
랭크: 2행 1, 열 2의 원소를 999로 설정합니다(인덱싱 방식은 아래를 참고하세요).
g2[1, 2] = 999
g2array([[ 0, 1, 2, 3, 4, 5],
[ 6, 7, 999, 9, 10, 11],
[ 12, 13, 14, 15, 16, 17],
[ 18, 19, 20, 21, 22, 23]])이에 상응하는 g의 원소도 수정됩니다.
garray([[[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[999, 9, 10, 11]],
[[ 12, 13, 14, 15],
[ 16, 17, 18, 19],
[ 20, 21, 22, 23]]])7.3 ravel
마지막으로 ravel 함수는 동일한 데이터를 가리키는 새로운 1차원 ndarray를 반환합니다:
g.ravel()array([ 0, 1, 2, 3, 4, 5, 6, 7, 999, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23])8 산술 연산
일반적인 산술 연산자(+, -, *, /, //, ** 등)는 모두 ndarray와 사용할 수 있습니다. 이 연산자는 원소별로 적용됩니다:
a = np.array([14, 23, 32, 41])
b = np.array([5, 4, 3, 2])
print("a + b =", a + b)
print("a - b =", a - b)
print("a * b =", a * b)
print("a / b =", a / b)
print("a // b =", a // b) # 몫
print("a % b =", a % b) # 나머지
print("a ** b =", a ** b)a + b = [19 27 35 43]
a - b = [ 9 19 29 39]
a * b = [70 92 96 82]
a / b = [ 2.8 5.75 10.66666667 20.5 ]
a // b = [ 2 5 10 20]
a % b = [4 3 2 1]
a ** b = [537824 279841 32768 1681]여기 곱셈은 행렬 곱셈이 아닙니다. 행렬 연산은 아래에서 설명합니다.
배열의 크기는 같아야 합니다. 그렇지 않으면 넘파이가 브로드캐스팅 규칙을 적용합니다.
9 브로드캐스팅
일반적으로 넘파이는 동일한 크기의 배열을 기대합니다. 그렇지 않은 상황에는 브로드캐시틍 규칙을 적용합니다:
9.1 규칙 1
배열의 랭크가 동일하지 않으면 랭크가 맞을 때까지 랭크가 작은 배열 앞에 1을 추가합니다.
h = np.arange(5).reshape(1,1, 5)
harray([[[0, 1, 2, 3, 4]]])여기에 (1,1,5) 크기의 3D 배열에 (5,) 크기의 1D 배열을 더해 보죠. 브로드캐스팅의 규칙 1이 적용됩니다!
h + [10, 20, 30, 40, 50] # 다음과 동일합니다: h + [[[10, 20, 30, 40, 50]]]array([[[10, 21, 32, 43, 54]]])9.2 규칙 2
특정 차원이 1인 배열은 그 차원에서 크기가 가장 큰 배열의 크기에 맞춰 동작합니다. 배열의 원소가 차원을 따라 반복됩니다.
k = np.arange(6).reshape(2, 3)
karray([[0, 1, 2],
[3, 4, 5]])(2,3) 크기의 2D ndarray에 (2,1) 크기의 2D 배열을 더해 보죠. 넘파이는 브로드캐스팅 규칙 2를 적용합니다:
k + [[100], [200]] # 다음과 같습니다: k + [[100, 100, 100], [200, 200, 200]]array([[100, 101, 102],
[203, 204, 205]])규칙 1과 2를 합치면 다음과 같이 동작합니다:
k + [100, 200, 300] # 규칙 1 적용: [[100, 200, 300]], 규칙 2 적용: [[100, 200, 300], [100, 200, 300]]array([[100, 201, 302],
[103, 204, 305]])또 매우 간단히 다음 처럼 해도 됩니다:
k + 1000 # 다음과 같습니다: k + [[1000, 1000, 1000], [1000, 1000, 1000]]array([[1000, 1001, 1002],
[1003, 1004, 1005]])9.3 규칙 3
규칙 1 & 2을 적용했을 때 모든 배열의 크기가 맞아야 합니다.
### 차원(배열 크기)가 달라서 생기는 문제
try:
k + [33, 44]
except ValueError as e:
print(e)operands could not be broadcast together with shapes (2,3) (2,) 브로드캐스팅 규칙은 산술 연산 뿐만 아니라 넘파이 연산에서 많이 사용됩니다. 아래에서 더 보도록 하죠. 브로드캐스팅에 관한 더 자세한 정보는 온라인 문서를 참고하세요.
10 업캐스팅
10.1 스킵
dtype이 다른 배열을 합칠 때 넘파이는 (실제 값에 상관없이) 모든 값을 다룰 수 있는 타입으로 업캐스팅합니다.
k1 = np.arange(5, dtype=np.uint8)
print(k1.dtype, k1)uint8 [0 1 2 3 4]k2 = k1 + np.array([5, 6, 7, 8, 9], dtype=np.int8)
print(k2.dtype, k2)int16 [ 5 7 9 11 13]모든 int8과 uint8 값(-128에서 255까지)을 표현하기 위해 int16이 필요합니다. 이 코드에서는 uint8이면 충분하지만 업캐스팅되었습니다.
k3 = k1 + 1.5
print(k3.dtype, k3)float64 [1.5 2.5 3.5 4.5 5.5]11 조건 연산자
조건 연산자도 원소별로 적용됩니다:
m = np.array([20, -5, 30, 40])
m < [15, 16, 35, 36]array([False, True, True, False])브로드캐스팅을 사용합니다:
m < 25 # m < [25, 25, 25, 25] 와 동일array([ True, True, False, False])불리언 인덱싱과 함께 사용하면 아주 유용합니다(아래에서 설명하겠습니다).
m[m < 25]array([20, -5])12 수학 함수와 통계 함수
ndarray에서 사용할 수 있는 수학 함수와 통계 함수가 많습니다.
12.1 ndarray 메서드
일부 함수는 ndarray 메서드로 제공됩니다. 예를 들면:
a = np.array([[-2.5, 3.1, 7], [10, 11, 12]])
print(a)
print("평균 =", a.mean())
# 축에 따라서 다르게 : axis= n[[-2.5 3.1 7. ]
[10. 11. 12. ]]
평균 = 6.766666666666667이 명령은 크기에 상관없이 ndarray에 있는 모든 원소의 평균을 계산합니다.
다음은 유용한 ndarray 메서드입니다:
for func in (a.min, a.max, a.sum, a.prod, a.std, a.var):
print(func.__name__, "=", func())min = -2.5
max = 12.0
sum = 40.6
prod = -71610.0
std = 5.084835843520964
var = 25.855555555555554이 함수들은 선택적으로 매개변수 axis를 사용합니다. 지정된 축을 따라 원소에 연산을 적용하는데 사용합니다. 예를 들면:
c=np.arange(24).reshape(2,3,4)
carray([[[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11]],
[[12, 13, 14, 15],
[16, 17, 18, 19],
[20, 21, 22, 23]]])c.sum(axis=0) # 첫 번째 축을 따라 더함, 결과는 3x4 배열array([[12, 14, 16, 18],
[20, 22, 24, 26],
[28, 30, 32, 34]])c.sum(axis=1) # 두 번째 축을 따라 더함, 결과는 2x4 배열array([[12, 15, 18, 21],
[48, 51, 54, 57]])c.sum(axis=2)array([[ 6, 22, 38],
[54, 70, 86]])여러 축에 대해서 더할 수도 있습니다:
c.sum(axis=(0,2)) # 첫 번째 축과 세 번째 축을 따라 더함, 결과는 (3,) 배열array([ 60, 92, 124])0+1+2+3 + 12+13+14+15, 4+5+6+7 + 16+17+18+19, 8+9+10+11 + 20+21+22+23(60, 92, 124)13 일반 함수
넘파이는 일반 함수(universal function) 또는 ufunc라고 부르는 원소별 함수를 제공합니다. 예를 들면 square 함수는 원본 ndarray를 복사하여 각 원소를 제곱한 새로운 ndarray 객체를 반환합니다:
a = np.array([[-2.5, 3.1, 7], [10, 11, 12]])
np.square(a)array([[ 6.25, 9.61, 49. ],
[100. , 121. , 144. ]])다음은 유용한 단항 일반 함수들입니다:
print("원본 ndarray")
print(a)
for func in (np.abs, np.sqrt, np.exp, np.log, np.sign, np.ceil, np.modf, np.isnan, np.cos):
print("\n", func.__name__)
print(func(a))원본 ndarray
[[-2.5 3.1 7. ]
[10. 11. 12. ]]
absolute
[[ 2.5 3.1 7. ]
[10. 11. 12. ]]
sqrt
[[ nan 1.76068169 2.64575131]
[3.16227766 3.31662479 3.46410162]]
exp
[[8.20849986e-02 2.21979513e+01 1.09663316e+03]
[2.20264658e+04 5.98741417e+04 1.62754791e+05]]
log
[[ nan 1.13140211 1.94591015]
[2.30258509 2.39789527 2.48490665]]
sign
[[-1. 1. 1.]
[ 1. 1. 1.]]
ceil
[[-2. 4. 7.]
[10. 11. 12.]]
modf
(array([[-0.5, 0.1, 0. ],
[ 0. , 0. , 0. ]]), array([[-2., 3., 7.],
[10., 11., 12.]]))
isnan
[[False False False]
[False False False]]
cos
[[-0.80114362 -0.99913515 0.75390225]
[-0.83907153 0.0044257 0.84385396]]
C:\Users\seong taek\AppData\Local\Temp\ipykernel_8044\4103705789.py:5: RuntimeWarning: invalid value encountered in sqrt
print(func(a))
C:\Users\seong taek\AppData\Local\Temp\ipykernel_8044\4103705789.py:5: RuntimeWarning: invalid value encountered in log
print(func(a))14 이항 일반 함수
두 개의 ndarray에 원소별로 적용되는 이항 함수도 많습니다. 두 배열이 동일한 크기가 아니면 브로드캐스팅 규칙이 적용됩니다:
a = np.array([1, -2, 3, 4])
b = np.array([2, 8, -1, 7])
np.add(a, b) # a + b 와 동일array([ 3, 6, 2, 11])np.greater(a, b) # a > b 와 동일array([False, False, True, False])np.maximum(a, b)array([2, 8, 3, 7])np.copysign(a, b)array([ 1., 2., -3., 4.])15 배열 인덱싱
15.1 1차원 배열
1차원 넘파이 배열은 보통의 파이썬 배열과 비슷하게 사용할 수 있습니다:
a = np.array([1, 5, 3, 19, 13, 7, 3])
a[3]19a[2:5]array([ 3, 19, 13])a[2:-1]array([ 3, 19, 13, 7])a[:2]array([1, 5])a[2::2]array([ 3, 13, 3])a[::-1]array([ 3, 7, 13, 19, 3, 5, 1])물론 원소를 수정할 수 있죠:
a[3]=999
aarray([ 1, 5, 3, 999, 13, 7, 3])슬라이싱을 사용해 ndarray를 수정할 수 있습니다:
a[2:5] = [997, 998, 999]
aarray([ 1, 5, 997, 998, 999, 7, 3])16 보통의 파이썬 배열과 차이점
보통의 파이썬 배열과 대조적으로 ndarray 슬라이싱에 하나의 값을 할당하면 슬라이싱 전체에 복사됩니다. 위에서 언급한 브로드캐스팅 덕택입니다.
a[2:5] = -1
aarray([ 1, 5, -1, -1, -1, 7, 3])또한 이런 식으로 ndarray 크기를 늘리거나 줄일 수 없습니다:
try:
a[2:5] = [1,2,3,4,5,6] # 너무 길어요
except ValueError as e:
print(e)could not broadcast input array from shape (6,) into shape (3,)원소를 삭제할 수도 없습니다:
try:
del a[2:5]
except ValueError as e:
print(e)cannot delete array elements중요한 점은 ndarray의 슬라이싱은 같은 데이터 버퍼를 바라보는 뷰(view)입니다. 슬라이싱된 객체를 수정하면 실제 원본 ndarray가 수정됩니다!
a_slice = a[2:6]
print(a_slice)
a_slice[1] = 1000
print(a_slice)
print(a) # 원본 배열이 수정됩니다![-1 -1 -1 7]
[ -1 1000 -1 7]
[ 1 5 -1 1000 -1 7 3]a[3] = 2000
a_slice # 비슷하게 원본 배열을 수정하면 슬라이싱 객체에도 반영됩니다!array([ -1, 2000, -1, 7])데이터를 복사하려면 copy 메서드를 사용해야 합니다:
print(a)
another_slice = a[2:6].copy()
another_slice[1] = 3000
print(a) # 원본 배열이 수정되지 않습니다[ 1 5 -1 2000 -1 7 3]
[ 1 5 -1 2000 -1 7 3]a[3] = 4000
another_slice # 마찬가지로 원본 배열을 수정해도 복사된 배열은 바뀌지 않습니다array([ -1, 3000, -1, 7])17 다차원 배열
다차원 배열은 비슷한 방식으로 각 축을 따라 인덱싱 또는 슬라이싱해서 사용합니다. 콤마로 구분합니다:
b = np.arange(48).reshape(4, 12)
barray([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11],
[12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23],
[24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35],
[36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47]])b[1, 2] # 행 1, 열 214b[1, :] # 행 1, 모든 열array([12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23])b[1, :].shape(12,)b[:, 1] # 모든 행, 열 1array([ 1, 13, 25, 37])주의: 다음 두 표현에는 미묘한 차이가 있습니다:
b[1, :]array([12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23])b[1:2, :]array([[12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23]])b[1, :].shape(12,)b[1:2, :].shape(1, 12)첫 번째 표현식은 (12,) 크기인 1D 배열로 행이 하나입니다. 두 번째는 (1, 12) 크기인 2D 배열로 같은 행을 반환합니다.
18 팬시 인덱싱(Fancy indexing)
관심 대상의 인덱스 리스트를 지정할 수도 있습니다. 이를 팬시 인덱싱이라고 부릅니다.
barray([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11],
[12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23],
[24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35],
[36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47]])b[(0,2), 2:5] # 행 0과 2, 열 2에서 4(5-1)까지array([[ 2, 3, 4],
[26, 27, 28]])b[:, (-1, 2, -1)] # 모든 행, 열 -1 (마지막), 2와 -1 (다시 반대 방향으로)array([[11, 2, 11],
[23, 14, 23],
[35, 26, 35],
[47, 38, 47]])여러 개의 인덱스 리스트를 지정하면 인덱스에 맞는 값이 포함된 1D ndarray를 반환됩니다.
b[(-1, 2, -1, 2), (5, 9, 1, 9)] # returns a 1D array with b[-1, 5], b[2, 9], b[-1, 1] and b[2, 9] (again)array([41, 33, 37, 33])18.1 Quiz
array([[24, 25], [36, 37]])
출력해보기
b[(2,3),0:2], b[2:4,0:2](array([[24, 25],
[36, 37]]),
array([[24, 25],
[36, 37]]))18.2 Quiz
- 2차원 배열 ’array_2d’에서 첫번째 행의 모든 요소 선택
- 2차원 배열 ’array_2d’에서 두번째 열의 모든 요소 선택
- 2차원 배열 ’array_2d’에서 25,30,40,45 선택
array_2d = np.array([[5, 10, 15],
[20, 25, 30],
[35, 40, 45]])
array_2darray([[ 5, 10, 15],
[20, 25, 30],
[35, 40, 45]])# 1
print(array_2d[0,:])
# 2
print(array_2d[:,1])
# 3
print(array_2d[1:3,1:3])[ 5 10 15]
[10 25 40]
[[25 30]
[40 45]]# 또는
array_2d[0,:],array_2d[:,1], array_2d[(1,2),1:3](array([ 5, 10, 15]),
array([10, 25, 40]),
array([[25, 30],
[40, 45]]))19 고차원
고차원에서도 동일한 방식이 적용됩니다. 몇 가지 예를 살펴 보겠습니다:
c = b.reshape(4,2,6)
carray([[[ 0, 1, 2, 3, 4, 5],
[ 6, 7, 8, 9, 10, 11]],
[[12, 13, 14, 15, 16, 17],
[18, 19, 20, 21, 22, 23]],
[[24, 25, 26, 27, 28, 29],
[30, 31, 32, 33, 34, 35]],
[[36, 37, 38, 39, 40, 41],
[42, 43, 44, 45, 46, 47]]])c[2, 1, 4] # 행렬 2, 행 1, 열 434c[2, :, 3] # 행렬 2, 모든 행, 열 3array([27, 33])어떤 축에 대한 인덱스를 지정하지 않으면 이 축의 모든 원소가 반환됩니다:
c[2, 1] # 행렬 2, 행 1, 모든 열이 반환됩니다. c[2, 1, :]와 동일합니다.array([30, 31, 32, 33, 34, 35])20 생략 부호 (...)
생략 부호(...)를 쓰면 모든 지정하지 않은 축의 원소를 포함합니다.
c[2, ...] # 행렬 2, 모든 행, 모든 열. c[2, :, :]와 동일array([[24, 25, 26, 27, 28, 29],
[30, 31, 32, 33, 34, 35]])c[2, 1, ...] # 행렬 2, 행 1, 모든 열. c[2, 1, :]와 동일array([30, 31, 32, 33, 34, 35])c[2, ..., 3] # 행렬 2, 모든 행, 열 3. c[2, :, 3]와 동일array([27, 33])c[..., 3] # 모든 행렬, 모든 행, 열 3. c[:, :, 3]와 동일array([[ 3, 9],
[15, 21],
[27, 33],
[39, 45]])21 불리언 인덱싱
불리언 값을 가진 ndarray를 사용해 축의 인덱스를 지정할 수 있습니다.
b = np.arange(48).reshape(4, 12)
barray([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11],
[12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23],
[24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35],
[36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47]])rows_on = np.array([True, False, True, False])
b[rows_on, :] # True에 해당하는 값 가져오기, 행 0과 2, 모든 열. b[(0, 2), :]와 동일array([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11],
[24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35]])cols_on = np.array([False, True, False] * 4)
b[:, cols_on] # 모든 행, 열 1, 4, 7, 10array([[ 1, 4, 7, 10],
[13, 16, 19, 22],
[25, 28, 31, 34],
[37, 40, 43, 46]])22 np.ix_
여러 축에 걸쳐서는 불리언 인덱싱을 사용할 수 없고 ix_ 함수를 사용합니다:
b[np.ix_(rows_on, cols_on)]array([[ 1, 4, 7, 10],
[25, 28, 31, 34]])np.ix_(rows_on, cols_on) # 추출 위치 출력(array([[0],
[2]], dtype=int64),
array([[ 1, 4, 7, 10]], dtype=int64))ndarray와 같은 크기의 불리언 배열을 사용하면 해당 위치가 True인 모든 원소를 담은 1D 배열이 반환됩니다. 일반적으로 조건 연산자와 함께 사용합니다:
b[b % 3 == 1]array([ 1, 4, 7, 10, 13, 16, 19, 22, 25, 28, 31, 34, 37, 40, 43, 46])23 반복
ndarray를 반복하는 것은 일반적인 파이썬 배열을 반복한는 것과 매우 유사합니다. 다차원 배열을 반복하면 첫 번째 축에 대해서 수행됩니다.
c = np.arange(24).reshape(2, 3, 4) # 3D 배열 (두 개의 3x4 행렬로 구성됨)
carray([[[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11]],
[[12, 13, 14, 15],
[16, 17, 18, 19],
[20, 21, 22, 23]]])for m in c:
print("아이템:")
print(m)아이템:
[[ 0 1 2 3]
[ 4 5 6 7]
[ 8 9 10 11]]
아이템:
[[12 13 14 15]
[16 17 18 19]
[20 21 22 23]]for i in range(len(c)): # len(c) == c.shape[0]
print("아이템:")
print(c[i])아이템:
[[ 0 1 2 3]
[ 4 5 6 7]
[ 8 9 10 11]]
아이템:
[[12 13 14 15]
[16 17 18 19]
[20 21 22 23]]ndarray에 있는 모든 원소를 반복하려면 flat 속성을 사용합니다:
for i in c.flat:
print("아이템:", i)아이템: 0
아이템: 1
아이템: 2
아이템: 3
아이템: 4
아이템: 5
아이템: 6
아이템: 7
아이템: 8
아이템: 9
아이템: 10
아이템: 11
아이템: 12
아이템: 13
아이템: 14
아이템: 15
아이템: 16
아이템: 17
아이템: 18
아이템: 19
아이템: 20
아이템: 21
아이템: 22
아이템: 2324 배열 쌓기
종종 다른 배열을 쌓아야 할 때가 있습니다. 넘파이는 이를 위해 몇 개의 함수를 제공합니다. 먼저 배열 몇 개를 만들어 보죠.
q1 = np.full((3,4), 1.0)
q1
# axisarray([[1., 1., 1., 1.],
[1., 1., 1., 1.],
[1., 1., 1., 1.]])q2 = np.full((4,4), 2.0)
q2array([[2., 2., 2., 2.],
[2., 2., 2., 2.],
[2., 2., 2., 2.],
[2., 2., 2., 2.]])q3 = np.full((3,4), 3.0)
q3array([[3., 3., 3., 3.],
[3., 3., 3., 3.],
[3., 3., 3., 3.]])24.1 vstack
vstack 함수를 사용하여 수직으로 쌓아보죠:
q4 = np.vstack((q1, q2, q3))
q4array([[1., 1., 1., 1.],
[1., 1., 1., 1.],
[1., 1., 1., 1.],
[2., 2., 2., 2.],
[2., 2., 2., 2.],
[2., 2., 2., 2.],
[2., 2., 2., 2.],
[3., 3., 3., 3.],
[3., 3., 3., 3.],
[3., 3., 3., 3.]])q4.shape(10, 4)q1, q2, q3가 모두 같은 크기이므로 가능합니다(수직으로 쌓기 때문에 수직 축은 크기가 달라도 됩니다).
24.2 hstack
hstack을 사용해 수평으로도 쌓을 수 있습니다:
q5 = np.hstack((q1, q3))
q5array([[1., 1., 1., 1., 3., 3., 3., 3.],
[1., 1., 1., 1., 3., 3., 3., 3.],
[1., 1., 1., 1., 3., 3., 3., 3.]])# concatenate도 가능
np.concatenate((q1,q3),axis=1)array([[1., 1., 1., 1., 3., 3., 3., 3.],
[1., 1., 1., 1., 3., 3., 3., 3.],
[1., 1., 1., 1., 3., 3., 3., 3.]])q5.shape(3, 8)q1과 q3가 모두 3개의 행을 가지고 있기 때문에 가능합니다. q2는 4개의 행을 가지고 있기 때문에 q1, q3와 수평으로 쌓을 수 없습니다:
try:
q5 = np.hstack((q1, q2, q3))
except ValueError as e:
print(e)all the input array dimensions for the concatenation axis must match exactly, but along dimension 0, the array at index 0 has size 3 and the array at index 1 has size 424.3 concatenate
concatenate 함수는 지정한 축으로도 배열을 쌓습니다.
q7 = np.concatenate((q1, q2, q3), axis=0) # vstack과 동일
q7array([[1., 1., 1., 1.],
[1., 1., 1., 1.],
[1., 1., 1., 1.],
[2., 2., 2., 2.],
[2., 2., 2., 2.],
[2., 2., 2., 2.],
[2., 2., 2., 2.],
[3., 3., 3., 3.],
[3., 3., 3., 3.],
[3., 3., 3., 3.]])q7.shape(10, 4)예상했겠지만 hstack은 axis=1으로 concatenate를 호출하는 것과 같습니다.
24.4 stack
stack 함수는 새로운 축을 따라 배열을 쌓습니다. 모든 배열은 같은 크기를 가져야 합니다.
q8 = np.stack((q1, q3))
q8array([[[1., 1., 1., 1.],
[1., 1., 1., 1.],
[1., 1., 1., 1.]],
[[3., 3., 3., 3.],
[3., 3., 3., 3.],
[3., 3., 3., 3.]]])q8.shape(2, 3, 4)25 배열 분할
분할은 쌓기의 반대입니다. 예를 들어 vsplit 함수는 행렬을 수직으로 분할합니다.
먼저 6x4 행렬을 만들어 보죠:
r = np.arange(24).reshape(6,4)
rarray([[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11],
[12, 13, 14, 15],
[16, 17, 18, 19],
[20, 21, 22, 23]])수직으로 동일한 크기로 나누어 보겠습니다:
### 3개의 변수로 분할 - 수직
r1, r2, r3 = np.vsplit(r, 3)
r1array([[0, 1, 2, 3],
[4, 5, 6, 7]])r2array([[ 8, 9, 10, 11],
[12, 13, 14, 15]])r3array([[16, 17, 18, 19],
[20, 21, 22, 23]])split 함수는 주어진 축을 따라 배열을 분할합니다. vsplit는 axis=0으로 split를 호출하는 것과 같습니다. hsplit 함수는 axis=1로 split를 호출하는 것과 같습니다:
### 2개의 변수로 분할 - 수평
r4, r5 = np.hsplit(r, 2)
r4array([[ 0, 1],
[ 4, 5],
[ 8, 9],
[12, 13],
[16, 17],
[20, 21]])r5array([[ 2, 3],
[ 6, 7],
[10, 11],
[14, 15],
[18, 19],
[22, 23]])26 배열 전치
읽어만 보기
transpose 메서드는 주어진 순서대로 축을 뒤바꾸어 ndarray 데이터에 대한 새로운 뷰를 만듭니다.
예를 위해 3D 배열을 만들어 보죠:
t = np.arange(24).reshape(4,2,3)
tarray([[[ 0, 1, 2],
[ 3, 4, 5]],
[[ 6, 7, 8],
[ 9, 10, 11]],
[[12, 13, 14],
[15, 16, 17]],
[[18, 19, 20],
[21, 22, 23]]])0, 1, 2(깊이, 높이, 너비) 축을 1, 2, 0 (깊이→너비, 높이→깊이, 너비→높이) 순서로 바꾼 ndarray를 만들어 보겠습니다:
t1 = t.transpose((1,2,0))
t1array([[[ 0, 6, 12, 18],
[ 1, 7, 13, 19],
[ 2, 8, 14, 20]],
[[ 3, 9, 15, 21],
[ 4, 10, 16, 22],
[ 5, 11, 17, 23]]])t1.shape(2, 3, 4)transpose 기본값은 차원의 순서를 역전시킵니다:
t2 = t.transpose() # t.transpose((2, 1, 0))와 동일
t2array([[[ 0, 6, 12, 18],
[ 3, 9, 15, 21]],
[[ 1, 7, 13, 19],
[ 4, 10, 16, 22]],
[[ 2, 8, 14, 20],
[ 5, 11, 17, 23]]])t2.shape(3, 2, 4)넘파이는 두 축을 바꾸는 swapaxes 함수를 제공합니다. 예를 들어 깊이와 높이를 뒤바꾸어 t의 새로운 뷰를 만들어 보죠:
t3 = t.swapaxes(0,1) # t.transpose((1, 0, 2))와 동일
t3array([[[ 0, 1, 2],
[ 6, 7, 8],
[12, 13, 14],
[18, 19, 20]],
[[ 3, 4, 5],
[ 9, 10, 11],
[15, 16, 17],
[21, 22, 23]]])t3.shape(2, 4, 3)27 선형 대수학
넘파이 2D 배열을 사용하면 파이썬에서 행렬을 효율적으로 표현할 수 있습니다. 주요 행렬 연산을 간단히 둘러 보겠습니다. 선형 대수학, 벡터와 행렬에 관한 자세한 내용은 Linear Algebra tutorial를 참고하세요.
28 행렬 전치
T 속성은 랭크가 2보다 크거나 같을 때 transpose()를 호출하는 것과 같습니다:
m1 = np.arange(10).reshape(2,5)
m1array([[0, 1, 2, 3, 4],
[5, 6, 7, 8, 9]])m1.Tarray([[0, 5],
[1, 6],
[2, 7],
[3, 8],
[4, 9]])T 속성은 랭크가 0이거나 1인 배열에는 아무런 영향을 미치지 않습니다:
m2 = np.arange(5)
m2array([0, 1, 2, 3, 4])m2.Tarray([0, 1, 2, 3, 4])먼저 1D 배열을 하나의 행이 있는 행렬(2D)로 바꾼다음 전치를 수행할 수 있습니다:
m2r = m2.reshape(1,5)
m2rarray([[0, 1, 2, 3, 4]])m2.shape, m2r.shape((5,), (1, 5))m2r.Tarray([[0],
[1],
[2],
[3],
[4]])29 행렬 곱셈
두 개의 행렬을 만들어 dot 메서드로 행렬 곱셈을 실행해 보죠.
n1 = np.arange(10).reshape(2, 5)
n1array([[0, 1, 2, 3, 4],
[5, 6, 7, 8, 9]])n2 = np.arange(15).reshape(5,3)
n2array([[ 0, 1, 2],
[ 3, 4, 5],
[ 6, 7, 8],
[ 9, 10, 11],
[12, 13, 14]])n1.dot(n2)array([[ 90, 100, 110],
[240, 275, 310]])주의: 앞서 언급한 것처럼 n1*n2는 행렬 곱셈이 아니라 원소별 곱셈(또는 아다마르 곱이라 부릅니다)입니다.
30 역행렬과 유사 역행렬
numpy.linalg 모듈 안에 많은 선형 대수 함수들이 있습니다. 특히 inv 함수는 정방 행렬의 역행렬을 계산합니다:
import numpy.linalg as linalg
m3 = np.array([[1,2,3],[5,7,11],[21,29,31]])
m3array([[ 1, 2, 3],
[ 5, 7, 11],
[21, 29, 31]])linalg.inv(m3)array([[-2.31818182, 0.56818182, 0.02272727],
[ 1.72727273, -0.72727273, 0.09090909],
[-0.04545455, 0.29545455, -0.06818182]])pinv 함수를 사용하여 유사 역행렬을 계산할 수도 있습니다:
linalg.pinv(m3)array([[-2.31818182, 0.56818182, 0.02272727],
[ 1.72727273, -0.72727273, 0.09090909],
[-0.04545455, 0.29545455, -0.06818182]])31 단위 행렬
행렬과 그 행렬의 역행렬을 곱하면 단위 행렬이 됩니다(작은 소숫점 오차가 있습니다):
m3.dot(linalg.inv(m3))array([[ 1.00000000e+00, -5.55111512e-17, 0.00000000e+00],
[-2.98372438e-16, 1.00000000e+00, -5.55111512e-17],
[ 5.78009862e-15, 1.27675648e-15, 1.00000000e+00]])eye 함수는 NxN 크기의 단위 행렬을 만듭니다:
np.eye(3)array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])32 QR 분해
qr 함수는 행렬을 QR 분해합니다:
q, r = linalg.qr(m3)
qarray([[-0.04627448, 0.98786672, 0.14824986],
[-0.23137241, 0.13377362, -0.96362411],
[-0.97176411, -0.07889213, 0.22237479]])rarray([[-21.61018278, -29.89331494, -32.80860727],
[ 0. , 0.62427688, 1.9894538 ],
[ 0. , 0. , -3.26149699]])q.dot(r) # q.r는 m3와 같습니다array([[ 1., 2., 3.],
[ 5., 7., 11.],
[21., 29., 31.]])33 행렬식
det 함수는 행렬식을 계산합니다:
linalg.det(m3) # 행렬식 계산43.9999999999999934 고윳값과 고유벡터
eig 함수는 정방 행렬의 고윳값과 고유벡터를 계산합니다:
eigenvalues, eigenvectors = linalg.eig(m3)
eigenvalues # λarray([42.26600592, -0.35798416, -2.90802176])eigenvectors # varray([[-0.08381182, -0.76283526, -0.18913107],
[-0.3075286 , 0.64133975, -0.6853186 ],
[-0.94784057, -0.08225377, 0.70325518]])m3.dot(eigenvectors) - eigenvalues * eigenvectors # m3.v - λ*v = 0array([[6.66133815e-15, 1.11022302e-16, 4.44089210e-16],
[7.10542736e-15, 2.88657986e-15, 3.10862447e-15],
[3.55271368e-14, 6.81746326e-15, 4.88498131e-15]])35 특잇값 분해
svd 함수는 행렬을 입력으로 받아 그 행렬의 특잇값 분해를 반환합니다:
m4 = np.array([[1,0,0,0,2], [0,0,3,0,0], [0,0,0,0,0], [0,2,0,0,0]])
m4array([[1, 0, 0, 0, 2],
[0, 0, 3, 0, 0],
[0, 0, 0, 0, 0],
[0, 2, 0, 0, 0]])U, S_diag, V = linalg.svd(m4)
Uarray([[ 0., 1., 0., 0.],
[ 1., 0., 0., 0.],
[ 0., 0., 0., -1.],
[ 0., 0., 1., 0.]])S_diagarray([3. , 2.23606798, 2. , 0. ])svd 함수는 Σ의 대각 원소 값만 반환합니다. 전체 Σ 행렬은 다음과 같이 만듭니다:
S = np.zeros((4, 5))
S[np.diag_indices(4)] = S_diag
S # Σarray([[3. , 0. , 0. , 0. , 0. ],
[0. , 2.23606798, 0. , 0. , 0. ],
[0. , 0. , 2. , 0. , 0. ],
[0. , 0. , 0. , 0. , 0. ]])Varray([[-0. , 0. , 1. , 0. , 0. ],
[ 0.4472136 , 0. , 0. , 0. , 0.89442719],
[-0. , 1. , 0. , 0. , 0. ],
[ 0. , 0. , 0. , 1. , 0. ],
[-0.89442719, 0. , 0. , 0. , 0.4472136 ]])U.dot(S).dot(V) # U.Σ.V == m4array([[1., 0., 0., 0., 2.],
[0., 0., 3., 0., 0.],
[0., 0., 0., 0., 0.],
[0., 2., 0., 0., 0.]])36 대각원소와 대각합
np.diag(m3) # m3의 대각 원소입니다(왼쪽 위에서 오른쪽 아래)array([ 1, 7, 31])np.trace(m3) # np.diag(m3).sum()와 같습니다3937 선형 방정식 풀기
solve 함수는 다음과 같은 선형 방정식을 풉니다:
- \(2x + 6y = 6\)
- \(5x + 3y = -9\)
coeffs = np.array([[2, 6], [5, 3]])
depvars = np.array([6, -9])
solution = linalg.solve(coeffs, depvars)
solutionarray([-3., 2.])solution을 확인해 보죠:
coeffs.dot(solution), depvars # 네 같네요(array([ 6., -9.]), array([ 6, -9]))좋습니다! 다른 방식으로도 solution을 확인해 보죠:
np.allclose(coeffs.dot(solution), depvars)True38 벡터화
한 번에 하나씩 개별 배열 원소에 대해 연산을 실행하는 대신 배열 연산을 사용하면 훨씬 효율적인 코드를 만들 수 있습니다. 이를 벡터화라고 합니다. 이를 사용하여 넘파이의 최적화된 성능을 활용할 수 있습니다.
예를 들어, \(sin(xy/40.5)\) 식을 기반으로 768x1024 크기 배열을 생성하려고 합니다. 중첩 반복문 안에 파이썬의 math 함수를 사용하는 것은 나쁜 방법입니다:
import math
data = np.empty((768, 1024))
for y in range(768):
for x in range(1024):
data[y, x] = math.sin(x*y/40.5) # 매우 비효율적입니다!작동은 하지만 순수한 파이썬 코드로 반복문이 진행되기 때문에 아주 비효율적입니다. 이 알고리즘을 벡터화해 보죠. 먼저 넘파이 meshgrid 함수로 좌표 벡터를 사용해 행렬을 만듭니다.
x_coords = np.arange(0, 1024) # [0, 1, 2, ..., 1023]
y_coords = np.arange(0, 768) # [0, 1, 2, ..., 767]
X, Y = np.meshgrid(x_coords, y_coords)
Xarray([[ 0, 1, 2, ..., 1021, 1022, 1023],
[ 0, 1, 2, ..., 1021, 1022, 1023],
[ 0, 1, 2, ..., 1021, 1022, 1023],
...,
[ 0, 1, 2, ..., 1021, 1022, 1023],
[ 0, 1, 2, ..., 1021, 1022, 1023],
[ 0, 1, 2, ..., 1021, 1022, 1023]])Yarray([[ 0, 0, 0, ..., 0, 0, 0],
[ 1, 1, 1, ..., 1, 1, 1],
[ 2, 2, 2, ..., 2, 2, 2],
...,
[765, 765, 765, ..., 765, 765, 765],
[766, 766, 766, ..., 766, 766, 766],
[767, 767, 767, ..., 767, 767, 767]])여기서 볼 수 있듯이 X와 Y 모두 768x1024 배열입니다. X에 있는 모든 값은 수평 좌표에 해당합니다. Y에 있는 모든 값은 수직 좌표에 해당합니다.
이제 간단히 배열 연산을 사용해 계산할 수 있습니다:
data = np.sin(X*Y/40.5)맷플롯립의 imshow 함수를 사용해 이 데이터를 그려보죠(matplotlib tutorial을 참조하세요).
import matplotlib.pyplot as plt
import matplotlib.cm as cm
fig = plt.figure(1, figsize=(7, 6))
plt.imshow(data, cmap=cm.hot)
plt.show()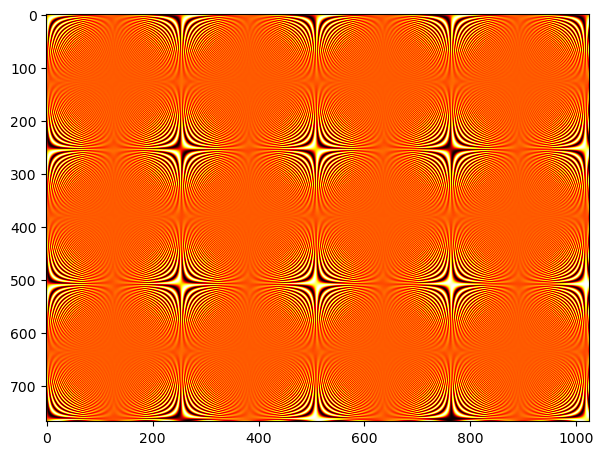
39 저장과 로딩
넘파이는 ndarray를 바이너리 또는 텍스트 포맷으로 손쉽게 저장하고 로드할 수 있습니다.
40 바이너리 .npy 포맷
랜덤 배열을 만들고 저장해 보죠.
a = np.random.rand(2,3)
aarray([[0.64010765, 0.96817264, 0.58635902],
[0.84834041, 0.25887928, 0.52484385]])np.save("my_array", a)끝입니다! 파일 이름의 확장자를 지정하지 않았기 때문에 넘파이는 자동으로 .npy를 붙입니다. 파일 내용을 확인해 보겠습니다:
with open("my_array.npy", "rb") as f:
content = f.read()
contentb"\x93NUMPY\x01\x00v\x00{'descr': '<f8', 'fortran_order': False, 'shape': (2, 3), } \n%u\x9e\x0b\xc3{\xe4?\n\\\x9e2E\xfb\xee?`,\xcc\xffs\xc3\xe2?\x90\x10F\xc8\x9a%\xeb?V\x0e-gz\x91\xd0?\x19y\xa1V\x85\xcb\xe0?"이 파일을 넘파이 배열로 로드하려면 load 함수를 사용합니다:
a_loaded = np.load("my_array.npy")
a_loadedarray([[0.64010765, 0.96817264, 0.58635902],
[0.84834041, 0.25887928, 0.52484385]])41 텍스트 포맷
배열을 텍스트 포맷으로 저장해 보죠:
np.savetxt("my_array.csv", a)파일 내용을 확인해 보겠습니다:
with open("my_array.csv", "rt") as f:
print(f.read())6.401076533253770018e-01 9.681726444858906877e-01 5.863590236707629799e-01
8.483404075236808950e-01 2.588792808130383483e-01 5.248438541418948278e-01이 파일은 탭으로 구분된 CSV 파일입니다. 다른 구분자를 지정할 수도 있습니다:
np.savetxt("my_array.csv", a, delimiter=",")이 파일을 로드하려면 loadtxt 함수를 사용합니다:
a_loaded = np.loadtxt("my_array.csv", delimiter=",")
a_loadedarray([[0.64010765, 0.96817264, 0.58635902],
[0.84834041, 0.25887928, 0.52484385]])42 압축된 .npz 포맷
여러 개의 배열을 압축된 한 파일로 저장하는 것도 가능합니다:
b = np.arange(24, dtype=np.uint8).reshape(2, 3, 4)
barray([[[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11]],
[[12, 13, 14, 15],
[16, 17, 18, 19],
[20, 21, 22, 23]]], dtype=uint8)np.savez("my_arrays", my_a=a, my_b=b)파일 내용을 확인해 보죠. .npz 파일 확장자가 자동으로 추가되었습니다.
with open("my_arrays.npz", "rb") as f:
content = f.read()
repr(content)[:180] + "[...]"'b"PK\\x03\\x04\\x14\\x00\\x00\\x00\\x00\\x00\\x00\\x00!\\x00D\\x08%/\\xb0\\x00\\x00\\x00\\xb0\\x00\\x00\\x00\\x08\\x00\\x14\\x00my_a.npy\\x01\\x00\\x10\\x00\\xb0\\x00\\x00\\x00\\x00\\x00\\x00\\x00\\xb0\\x00\\x00\\x00\\x00[...]'다음과 같이 이 파일을 로드할 수 있습니다:
my_arrays = np.load("my_arrays.npz")
my_arrays<numpy.lib.npyio.NpzFile at 0x2665066fd00>게으른 로딩을 수행하는 딕셔너리와 유사한 객체입니다:
my_arrays.keys()KeysView(<numpy.lib.npyio.NpzFile object at 0x000002665066FD00>)my_arrays["my_a"]array([[0.64010765, 0.96817264, 0.58635902],
[0.84834041, 0.25887928, 0.52484385]])43 그 다음은?
넘파이 기본 요소를 모두 배웠지만 훨씬 더 많은 기능이 있습니다. 이를 배우는 가장 좋은 방법은 넘파이를 직접 실습해 보고 훌륭한 넘파이 문서에서 필요한 함수와 기능을 찾아 보세요.